数据集与问题描述
数据集测量了56条欧洲河鲈(Perca fluviatilis)的六项指标
- Weight Weight of the fish (in g)
- Length1 Length from the nose to the beginning of the tail (in cm)
- Length2 Length from the nose to the notch of the tail (in cm)
- Length3 Length from the nose to the end of the tail (in cm)
- Height Maximal height (in cm)
- Width Maximal width (in cm)
我们想知道鱼的重量和其他指标的关系。
数据集可以在 fish.data下载
相关库的加载和数据的读入
1
2
3
4
5
6
7
8
| requirements <- c('car', 'lars', 'tidyr', 'GGally')
for (package in requirements) {
if (!require(package, character.only=T, quietly=T)) {
install.packages(package)
library(package, character.only=T)
}
}
fishData <- read.table('fish.data', header=T)
|
探索性数据分析
通常拿到一个问题首先要做的就是对数据的探索性分析。惨痛的教训告诉我们,这一步绝不应该是走流程。
1
2
3
4
5
6
7
8
9
10
11
12
13
| dim(fishData)
## [1] 56 6
head(fishData)
## Weight Length1 Length2 Length3 Height Width
## 1 5.9 7.5 8.4 8.8 2.1120 1.4080
## 2 32.0 12.5 13.7 14.7 3.5280 1.9992
## 3 40.0 13.8 15.0 16.0 3.8240 2.4320
## 4 51.5 15.0 16.2 17.2 4.5924 2.6316
## 5 70.0 15.7 17.4 18.5 4.5880 2.9415
## 6 100.0 16.2 18.0 19.2 5.2224 3.3216
ggpairs(fishData, pch=19, cex=.2) # see the picture below
n <- dim(fishData)[1] # 56
p <- dim(fishData)[2] # 6
|
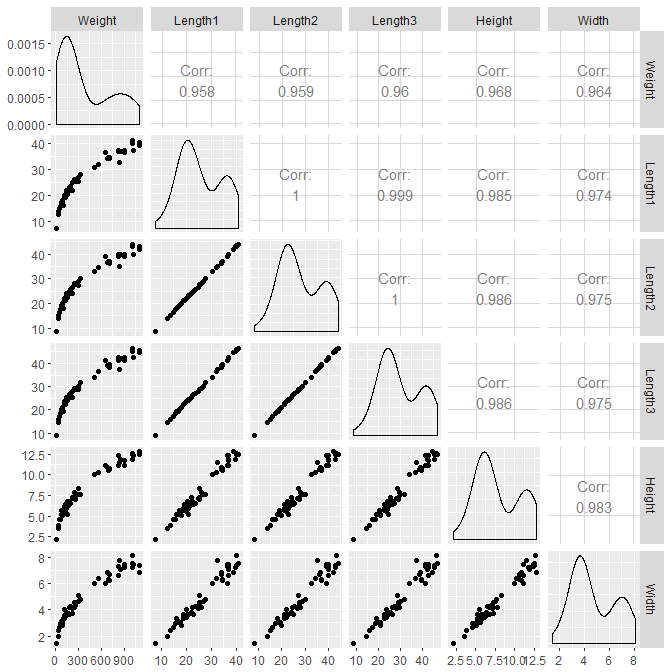
从图中可以清晰的看到两点
- 五个自变量间高度相关,这和预期完全一致
- 数据出现了双峰的分布,仔细观察可以看到数据间存在一段gap(事实上,这是我们后期才发现的特性,因为当时只是使用了
pairs验证了自变量的高度相关性,漏掉了这一值得注意的信息)
尝试建模
一个显然不合理的模型
我们先尝试将所有变量拿来预测,直观上其结果必定会十分糟糕,我们只是验证一下
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
| simpleModel <- lm(Weight~.,fishData)
summary(simpleModel)
## Call:
## lm(formula = Weight ~ ., data = fishData)
##
## Residuals:
## Min 1Q Median 3Q Max
## -125.03 -52.44 -26.28 34.73 301.66
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -556.59 60.66 -9.175 2.68e-12 ***
## Length1 -3.13 57.88 -0.054 0.9571
## Length2 -38.50 88.55 -0.435 0.6656
## Length3 42.92 60.09 0.714 0.4784
## Height 65.66 30.00 2.189 0.0333 *
## Width 64.90 36.77 1.765 0.0836 .
## ---
## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
##
## Residual standard error: 87.11 on 50 degrees of freedom
## Multiple R-squared: 0.9429, Adjusted R-squared: 0.9372
## F-statistic: 165.2 on 5 and 50 DF, p-value: < 2.2e-16
vif(simpleModel)
## Length1 Length2 Length3 Height Width
## 1780.07795 4626.41486 2376.77263 54.03761 30.85782
|
可以从VIF看到自变量间高度相关(三个长度的VIF更是达到了令人发指的四位数),而五个系数的方差都很大,基本没有什么解释性,这就是之前谈到的多重共线性的危害。
使用Lasso选择变量
我们知道由于Lasso可以起到选择变量的作用,因为其切点更有可能出现在坐标轴上。我们使用lars实现这一功能
1
2
3
4
| plot(lars(fishData[,-1] %>% data.matrix(), fishData[,1]))
vif(lm(Weight~Height+Width, fishData))
## Height Width
## 29.56642 29.56642
|
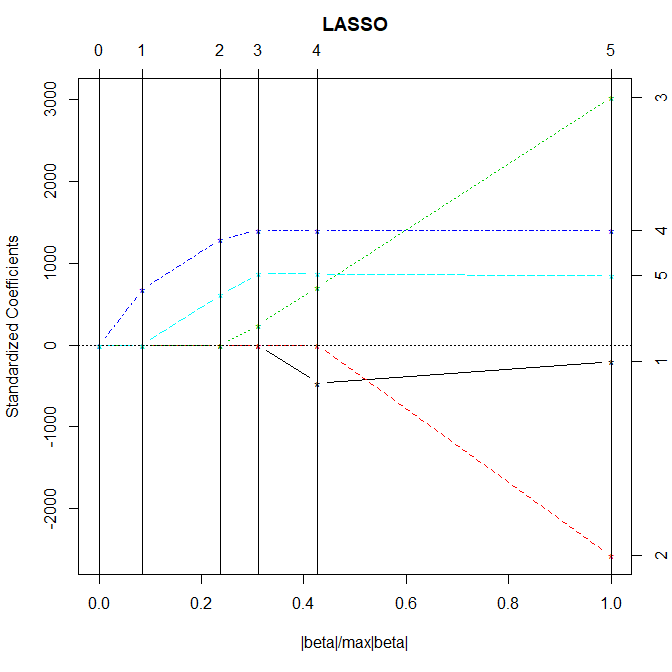
从图中可以看到,随着惩罚的逐渐降低,变量4和变量5依次加入到模型中,我们检查这样的模型的VIF,发现仍然高达30。因此即使是双变量都存在着严重的共线性问题,这从根本上断绝了多元回归的路子。
PCA降维
既然现在变量间高度相关,使用PCA提取出一个“综合体型”这样的想法是十分自然的。我们先使用PCA看下效果
1
2
| pca <- prcomp(fishData[,-1], center=F, scale=F)
screeplot(pca, type='lines')
|
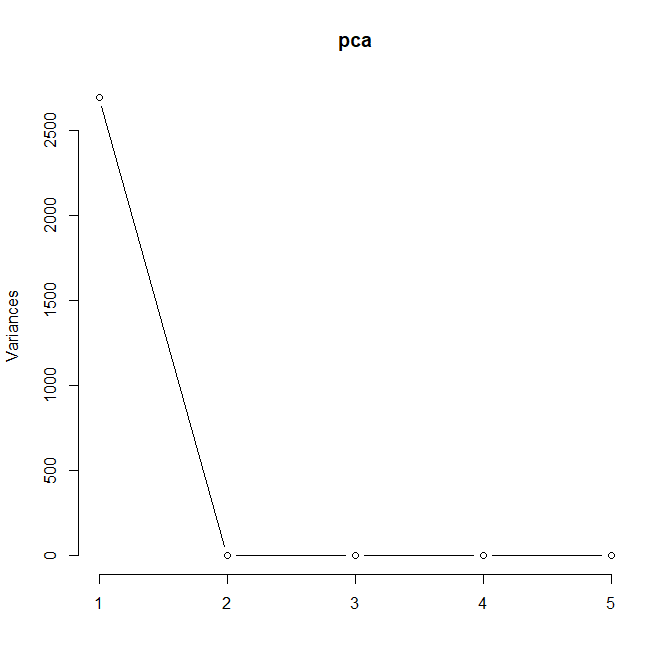
毫不意外的,第一主成分占走了绝大多数方差,因此我们只能选用第一主成分。然后我们通过散点图看下主成分和因变量之间的关系。
1
2
3
| y <- fishData[,1]
x <- -pca$x[,1]
plot(x, y, pch=19, cex=.2)
|
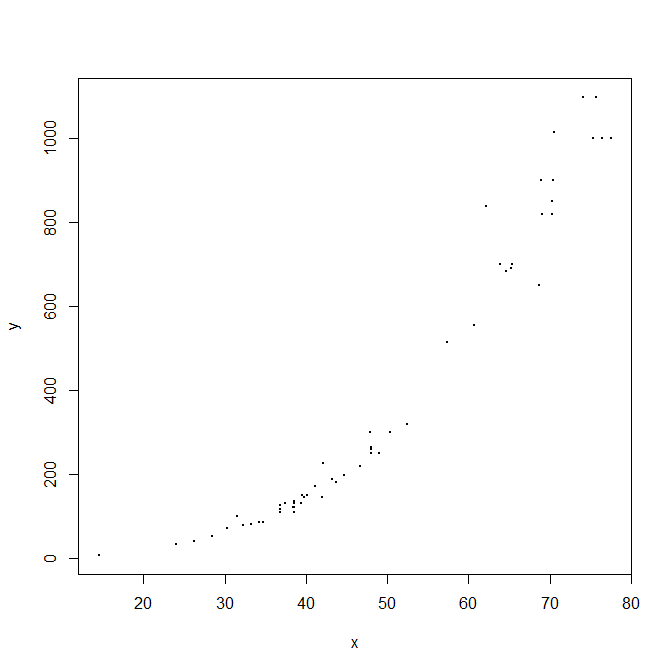
显然线性模型是不合适的,从直观上体重也不会随身高线性增长,相反他们应该有一个幂的关系。因此我们考虑对双放取对数后转化为线性模型来求解。
但是,现实中使用PCA是一个很麻烦的问题,你还得手动测量五个长度,然后根据一堆奇奇怪怪的系数(这个系数也不见得稳定,因为样本bias)换算得出。注意到其和Length是高度相关的,因此不如直接用某个长度更为方便直接。三个Length用哪个呢?个人更倾向于使用Length2,因为其测量相对简单:量头部到尾巴缺口的距离即可,不像尾巴的头部或尾部难以定位。因此后面我们都只使用Length2代替x。
对数化模型
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
| plot(y=log(y), x=log(x), pch=19, cex=.2)
log.fit <- lm(log(y)~log(x))
summary(log.fit)
## Call:
## lm(formula = log(y) ~ log(x))
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.21311 -0.08150 -0.00789 0.04987 0.38451
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -4.86097 0.15158 -32.07 <2e-16 ***
## log(x) 3.15296 0.04606 68.46 <2e-16 ***
## ---
## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
##
## Residual standard error: 0.1177 on 54 degrees of freedom
## Multiple R-squared: 0.9886, Adjusted R-squared: 0.9884
## F-statistic: 4686 on 1 and 54 DF, p-value: < 2.2e-16
|
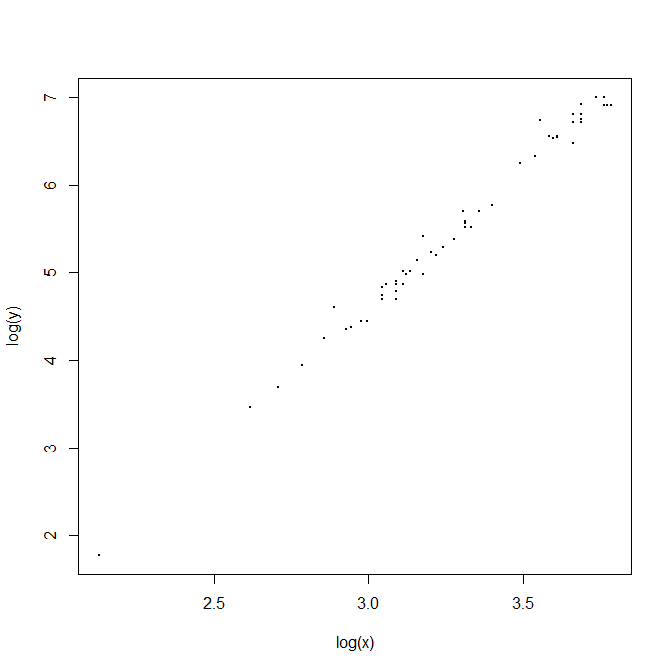
取对数后可以看到一个相当漂亮的线性关系,其拟合效果也相当不错,\(R^2\)达到了0.9886。事实上这个模型基本上可以收工了。但当观察到\(\log(x)\)的系数为\(3.15\)时,我们不禁有一个大胆的想法——如果用简单的三次方模型是否可行呢?
当然,冷静看一下标准差,不难看出\(3\)不太可能落在\(95\%\)的置信区间里。让我们用代码尝试一下
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
| summary(lm(log(y)-3*log(x)~log(x)))
## Call:
## lm(formula = log(y) - 3 * log(x) ~ log(x))
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.21311 -0.08150 -0.00789 0.04987 0.38451
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -4.86097 0.15158 -32.068 < 2e-16 ***
## log(x) 0.15296 0.04606 3.321 0.00161 **
## ---
## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
##
## Residual standard error: 0.1177 on 54 degrees of freedom
## Multiple R-squared: 0.1696, Adjusted R-squared: 0.1542
## F-statistic: 11.03 on 1 and 54 DF, p-value: 0.001613
|
\(\beta=3\)的p-value为0.00161,算是不太可能了。当然我们也可以argue说因为数据量太小可能有bias,因此我们就使用一个三次方模型使得模型更好解释,这就见仁见智了。
尝试更高阶的模型
之前我们得到了一个\(3.12\)阶的模型,然后发现降到\(3\)阶的想法不太被支持,但我们确实更喜欢整数次方的拟合结果,那么\(y=a_0+a_1x+a_2x^2+a_3x^3\)怎么样呢?
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
| x2 <- x^2
x3 <- x^3
summary(lm(y~x3+x2))
## Call:
## lm(formula = y ~ x3 + x2)
##
## Residuals:
## Min 1Q Median 3Q Max
## -154.792 -28.543 -3.992 18.650 249.137
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -60.410113 38.040283 -1.588 0.11822
## x3 0.009296 0.002942 3.160 0.00261 **
## x2 0.206276 0.139342 1.480 0.14470
## ---
## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
##
## Residual standard error: 60.65 on 53 degrees of freedom
## Multiple R-squared: 0.9707, Adjusted R-squared: 0.9696
## F-statistic: 876.9 on 2 and 53 DF, p-value: < 2.2e-16
summary(lm(y~x3+x))
## Call:
## lm(formula = y ~ x3 + x)
##
## Residuals:
## Min 1Q Median 3Q Max
## -152.74 -24.75 -4.65 18.25 254.74
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -76.144274 65.819272 -1.157 0.253
## x3 0.012291 0.001292 9.514 4.66e-13 ***
## x 3.840499 3.597097 1.068 0.291
## ---
## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
##
## Residual standard error: 61.24 on 53 degrees of freedom
## Multiple R-squared: 0.9701, Adjusted R-squared: 0.969
## F-statistic: 859.7 on 2 and 53 DF, p-value: < 2.2e-16
|
可以看到当\(x^3\)引入后,很难继续引入\(x\)与\(x^2\)。当然,其实这么直接粗暴的引入\(x\)和\(x^2\)是有问题的——会带来共线性问题。但这里我们只是做一些探索性的尝试,就不去标准化了。
数据标准化
当然,尝试下对数据标准化也不是不行,只是在这个数据上结果并没有显著改善,而且解释性也会变得很差(试想你要如何解释标准化的系数?只能说我们有一个“标准”的鱼,然后根据体型上对标准鱼的偏差计算出重量上对标准鱼的偏差,再换算会鱼的重量。因此这里只放相关的代码,有兴趣的读者自行尝试。
1
2
3
4
5
6
7
8
| nx1 <- scale(x)
nx2 <- nx1^2
nx3 <- nx1^3
ny <- scale(y)
plot(nx1, ny, pch=19, cex=.2)
nfit <- lm(ny~nx1)
step(nfit, scope=list(lower=ny~1, upper=ny~nx1+nx2+nx3))
summary(lm(ny~nx1+nx2))
|
阶段性成果
经过思考,我们最后还是无法抵挡整数次方带来的诱惑,因此采取\(y=a+bx^3\)这样的模型。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
| summary(lm(y~x3))
## Call:
## lm(formula = y ~ x3)
##
## Residuals:
## Min 1Q Median 3Q Max
## -153.502 -17.969 -5.177 9.488 262.964
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -7.1415073 12.4741568 -0.573 0.569
## x3 0.0136251 0.0003291 41.398 <2e-16 ***
## ---
## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
##
## Residual standard error: 61.32 on 54 degrees of freedom
## Multiple R-squared: 0.9695, Adjusted R-squared: 0.9689
## F-statistic: 1714 on 1 and 54 DF, p-value: < 2.2e-16
|
注意到,截距项的p-value达到了0.693,这意味着我们可以抛弃掉这一参数而得到更精炼的模型
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
| newModel <- lm(y~x3+0)
summary(newModel)
## Call:
## lm(formula = y ~ x3 + 0)
##
## Residuals:
## Min 1Q Median 3Q Max
## -149.801 -23.571 -11.324 9.543 261.914
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## x3 0.0134831 0.0002149 62.75 <2e-16 ***
## ---
## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
##
## Residual standard error: 60.94 on 55 degrees of freedom
## Multiple R-squared: 0.9862, Adjusted R-squared: 0.986
## F-statistic: 3938 on 1 and 55 DF, p-value: < 2.2e-16
plot(x3, y, xlab="x^3", pch=19, cex=.2)
lines(abline(newModel))
|
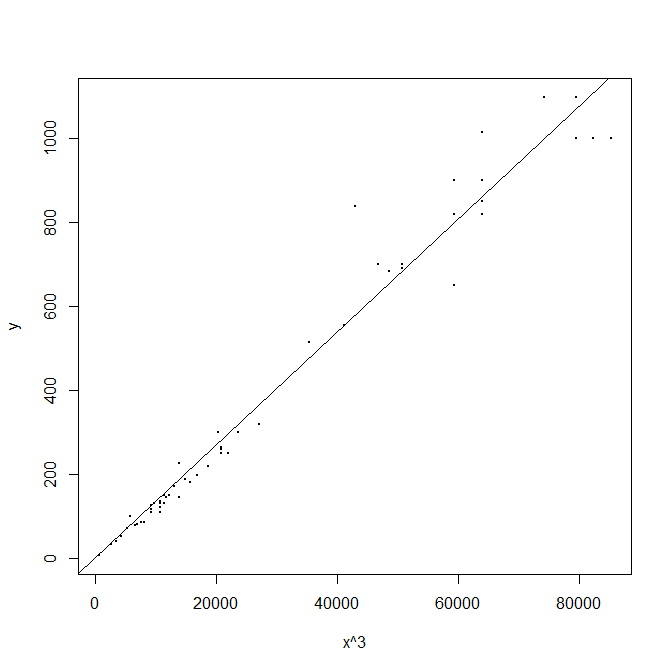
值得注意的时,使用\(\log(y)=3*\log(x)+b\)也能得到一个模型,这模型和上面直接三次方你和出来的模型是不一样的。选用哪种模型也是一个见人见智的事情。直接\(y=ax^3\)的\(\mathrm{SSE}\)肯定更小,但这样拟合的模型自变量相对不太好——如果\(x\)相对还比较均匀,那么\(x^3\)到后期可能就相当稀疏了,其误差带来的影响也会被放大,因此此时我们可能更喜欢对数化后的线性模型。
1
2
3
4
| mean(log(y)-3*log(x))
# [1] -4.360281
exp(mean(log(y)-3*log(x)))
# [1] 0.0127748
|
得到的模型为 \(y=0.01277 x^3\)。
在下面的讨论中,我们都使用对数化的模型做进一步的改进。当然读者也可以尝试用\(y=ax^3\)直接拟合然后尝试下面的改进。
还可以更好
上述这个模型基本上没有问题了,简洁明了有针对性,物理解释也足够好,过零点简直是一个意外的bonus。但当做模型诊断的时候,我们发现了这样的问题。
1
2
3
4
5
6
| par(mfrow=(c(2,2)))
plot(x3, newModel$residuals, xlab="x^3", ylab="e", pch=19, cex=.2)
plot(predict(newModel), newModel$residuals, xlab="yhat", ylab="e", pch=19, cex=.2)
plot(newModel$residuals[-n], newModel$residuals[-1], xlab="e_i", ylab="e_i+1", pch=19, cex=.2)
qqnorm(newModel$residuals, pch=19, cex=.2)
sum(newModel$residuals^2)/(n-1)
|
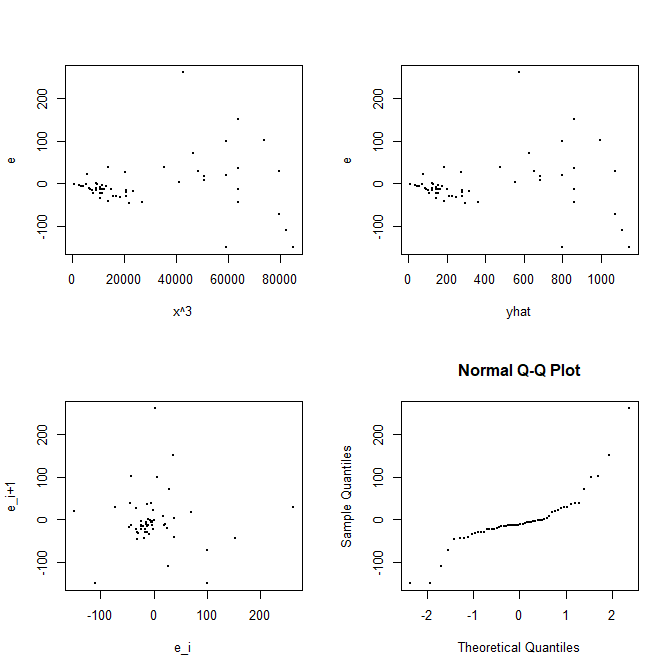
可以看到一个是误差的正态性不太好,有重尾的趋势。但事实上正态性假设在现实中许多时候是可有可无的。
另一个问题就是随着\(X\)或者\(\widehat{Y}\)的增大,误差也会随之增大,但这事实上是因为\(x^3\)放大了这一效应,在对数视角上看,其误差并没有明显的增大,还是可以接受的。
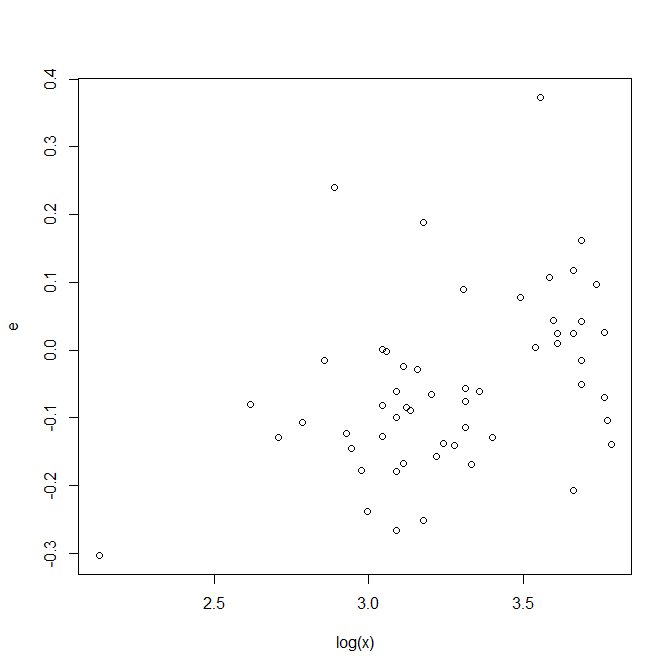
但我们能不能做一些改进呢?还记得最开始做探索性分析的时候我们发现的现象么?我们可以做这样的推想:这里面的鱼有幼年期和成熟期之分(或者是两个亚种?),因此体型有较大的差异。这可能导致他们三次方的系数不一样。因此,我们可以使用一个因子isBig标志其是否为大鱼,然后做一个协方差分析。
经过观察,数据的gap在Height比较大,因此我们选择在此维度作为划分,且取了gap中一个中庸的值9.0作为划分。此时,我们设想的模型是
\[Y=(a+b\cdot\mathrm{isBig})x^3\]
你可以使用两种方法得到相关的参数,一个简单的方法自然是分别回归两个模型,然后拼凑出对应的系数,即
1
2
3
4
5
6
7
| isBig <- fishData$Height > 9
small.coef <- mean(log(y[!isBig])-3*log(x[!isBig]))
big.coef <- mean(log(y[isBig])-3*log(x[isBig]))
exp(small.coef)
# [1] 0.01225077
exp(big.coef)
# [1] 0.01386048
|
当然另一个做法是直接使用回归的方式,这样做的好处是可以让R帮我们算一下\(t\)检验等。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
| final.model <- lm(log(y)-3*log(x)~isBig)
summary(final.model)
## Call:
## lm(formula = log(y) - 3 * log(x) ~ isBig)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.23500 -0.06384 -0.00319 0.04192 0.34607
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -4.40217 0.01884 -233.716 < 2e-16 ***
## isBigTRUE 0.12345 0.03234 3.818 0.000349 ***
## ---
## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
##
## Residual standard error: 0.1146 on 54 degrees of freedom
## Multiple R-squared: 0.2125, Adjusted R-squared: 0.198
## F-statistic: 14.58 on 1 and 54 DF, p-value: 0.0003491
|
可以看到这个因子的引入还是有用的。
至此,我们得到了我们使用的最终模型
\[\log(\mathrm{Weight})=(-4.40217+0.12345\mathrm{isBig}) + 3\log(\mathrm{Length2})\] \[\mathrm{Weight}=(0.0125+0.00161\mathrm{isBig}) (\mathrm{Length2})W^3\]
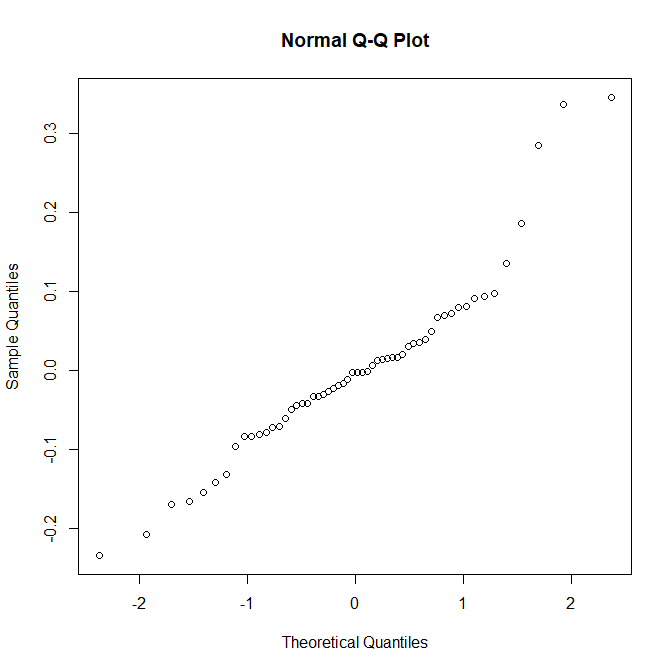
模型诊断
由于我们已经写死了三次幂的关系,因此一般的像检测\(X,\,Y\)的离群值意义相对不大,因此只做一些基础性的诊断分析。
1
| qqnorm(final.model$resuiduals)
|
至此,一个线性模型的分析任务就完成了,谢谢各位读者。








